肿瘤相关LncRNA编码肽段的数据库:
SPENCER | A comprehensive database for small peptides encoded by ncRNA in cancer patients:
http://spencer.renlab.org/#/home

背景数据集介绍
SPENCER主要利用蛋白质谱的方式来鉴定LncRNA相关的肽段。
首先,作者在 [[iProX_ProteomeXchange-蛋白质谱原始数据检索数据库|ProteomeXchange]] 当中检索和肿瘤有关的,最终得到来自55个数据集当中的1726个肿瘤样本的质谱数据。
同时,为了鉴定所有质谱得到的序列。作者首先从Uniprot当中获得了所有已知的蛋白序列,另外对于LncRNA而言,先在RNAcentral当中获得所有lncRNA的序列,首先使用 CPAT: https://cpat.readthedocs.io/en/latest/ 来筛选可以表达蛋白的LncRNA。同时使用ORFfinder: https://www.ncbi.nlm.nih.gov/orffinder/ 来预测LncRNA的开放阅读框,进一步把这些编码区域翻译成氨基酸序列。
利用以上的数据,作者使用MaxQuant来分析和寻找质谱当中的LncRNA编码的肽段。最终作者一共鉴定出29, 526个ncRNA相关的肽段。
最后对鉴定出来的肽段数据进行差异分析以及免疫原性分析就得到了整个SPENCER的所有的数据了。
数据库使用
SPENCER一共提供了三个主要的功能:1)数据库浏览;2)数据检索以及3)BLAST
浏览和检索
在数据的浏览当中,我们可以查看这个数据库的所有基本信息,比如有多少个肿瘤特异性的肽
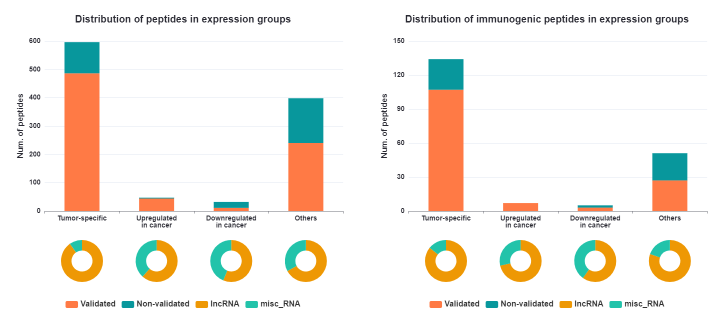
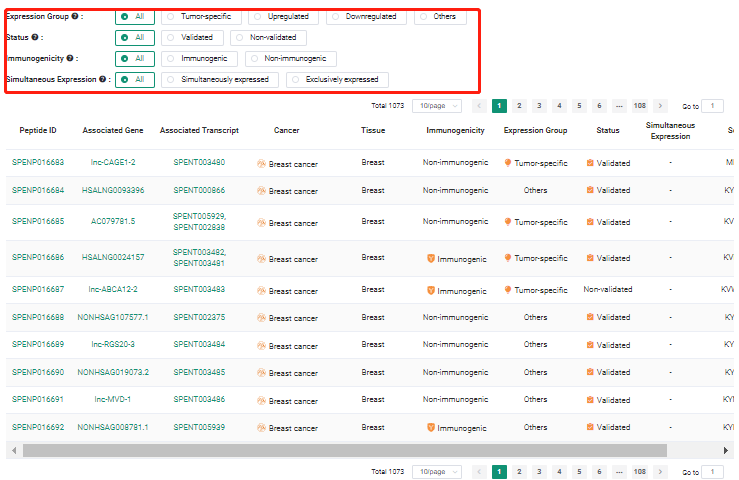
或者所有的数据表格。在里面也可以进行筛选是否是肿瘤特异性的,是否经过验证的以及是否有免疫原性。
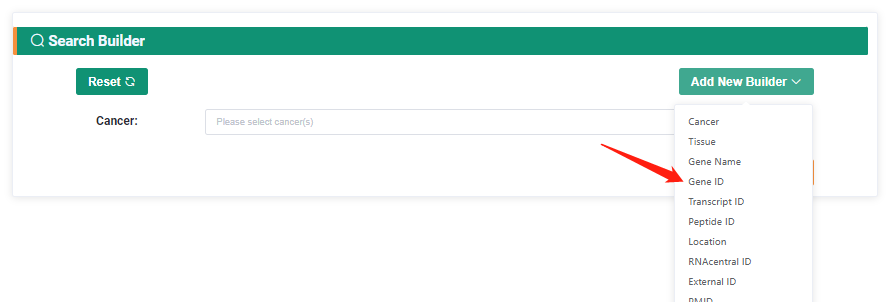
同时在检索当中,可以检索目标数据。最基本的检索是检索相关肿瘤数据,如果有想要进一步检索的条件,可以在Add New Builder当中进行添加。比如添加Gene ID
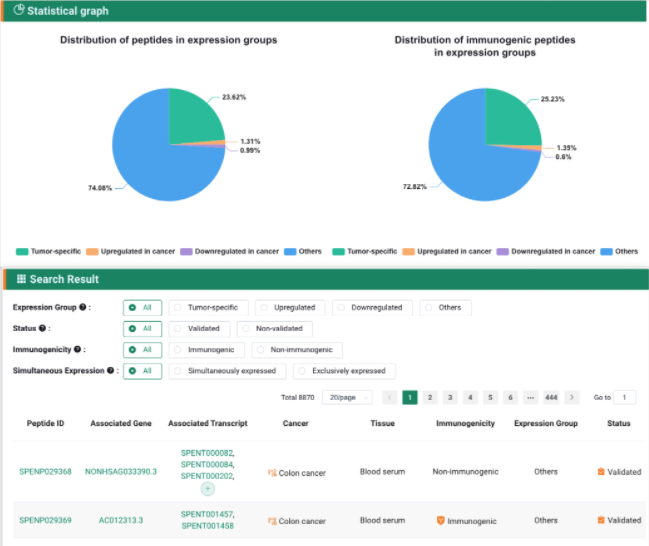
检索的结果主要是通过一个统计图和表格的形式来进行展示。
blast
除了一般数据库的功能之外,在SPENCER当中也提供了Blast的功能。方便使用lncRNA序列直接预测可以编码的肽段。其中序列的输入是以 [[Fasta基因序列格式]] 的格式输入。
以上就是SPENCER主要使用功能了,对于lncRNA的功能之前一直以ceRNA的功能。随着研究的深入如果一个LncRNA编码相关肽段也属于一个老的热点的新机制吧。如果有研究lncRNA的可以尝试的预测一下试试。
另外SPENCER主要是用来预测肿瘤相关肽的。如果要做其他疾病的话,其实参照其数据库的分析流程,可以直接检索其他疾病的进行分析即可。如果数据多了也可以构建一个数据库的。














